支持向量机(Support Vector Machine, SVM)是一类按监督学习(supervised learning)方式对数据进行广义线性分类,其决策边界是对学习样本求解的最大边距超平面(maximum-margin hyperplane)。SVM可以通过核函数进行非线性分类,是常见的核学习(kernel learning)方法之一。
什么是支持向量机
支持向量机(support vector machines)是寻找一个超平面来对样本进行分割,分割的原则是间隔最大化,最终转化为一个凸二次规划问题来求解。它既能解决线性可分又能解决线性不可能,既能解决分类问题又能完成回归问题。
当训练样本线性可分时使用硬间隔最大化(Hard Margin SVM)或者近似线性可分时使用软件最大化(Soft Margin SVM)。当训练样本线性不可分时使用核函数和软间隔最大化。
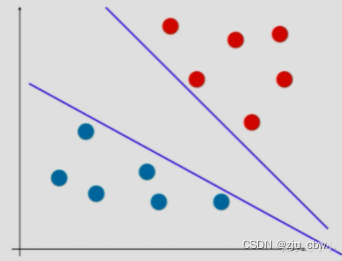
在实际问题中往往都存在着决策边界不唯一的情况,这就是不适定问题。给定训练样本集 D={(x1,y1),(x2,y2),…,(xm,ym)},yi∈{-1,1} 分类算法的基本思想就是基于训练集在样本空间中找到一个划分超平面,但是能将训练样本分开的划分超平面可能有很多,所以,应该努力地去找哪一个?
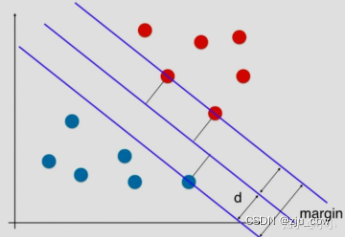
而svm找到的这条直线希望距离最近的红色的点和蓝色的点,距离决策边界尽可能的远,这样就能保证模型的泛化能力。svm尝试寻找一个最优的决策边界,距离两个类别最近的样本最远,图中3个点到决策边界距离相同。这三个点就叫做**支持向量(support vector)**。而平行于决策边界的两条直线之间的距离就是 margin,svm就是要最大化 margin ,这样就把这个问题转化称为最优化问题。
支持向量机背后的最优化问题

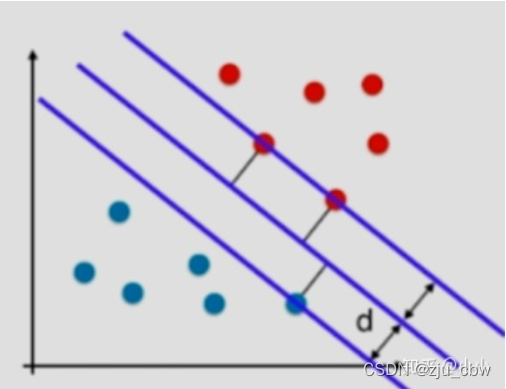
中间那根直线就是决策边界 wTx+b=0 ,而上下两根直线意味着距离一定大于 d,从而

此时,假设两类样本分别为1和-1。然后将上述式子的左右两侧同时除以 d ,得到

其中 ||w|| 和 d 都是一个数,此时消去分母,得到


Soft Margin和SVM正则化
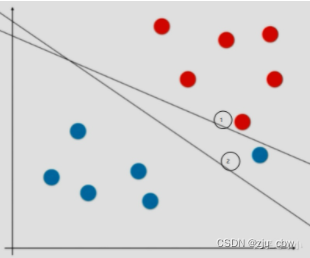
如果在实际应用过程中有一个蓝色的点出现在红色的点附近,即两类相对较为接近,但整体跟蓝色的点差异明显,可以看做是一个特殊点或者错误的奇点,此时就会误导,导致最终的hard margin分类边界是直线1,此时模型的泛化能力就值得怀疑。正常来说应该像直线2一样忽略那个极度特殊的蓝点,保证大多数的数据到直线的距离最远,可能这才是最好的分类边界,也就是泛化能力更高。这也能间接地说明如果模型的准确率过高可能会导致模型的过拟合。
还有一种更一般的例子,那就是如果有一个蓝色的点混进了红色的点当中,这就导致数据集根本就是线性不可分的情况,根本找不出一条直线能够将这两类分开,在这种情况下Hard margin就不再是泛化能力强不强的问题,而是根本找不出一条直线将其分开。
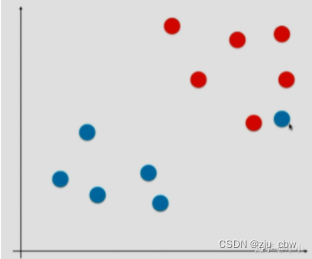
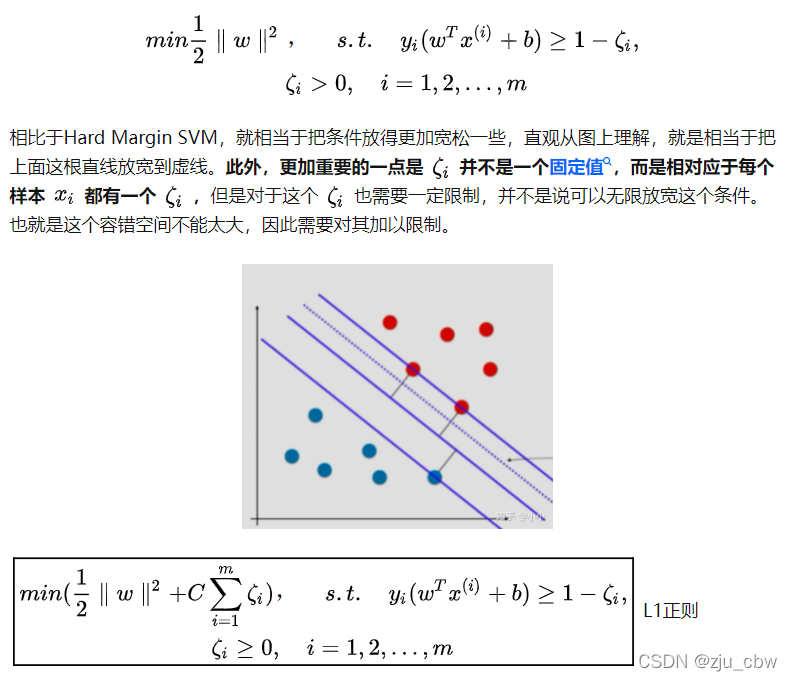
因此不管才以上两种情况的哪个角度出发,都应该考虑给予svm模型部分容错能力。由此引出Soft Margin SVM。

sklearn中的SVM
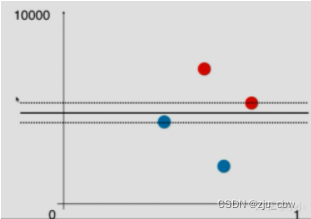
在实际使用SVM的时候和KNN一样需要对数据进行标准化处理,因为这两者都涉及距离。因为当数据尺度相差过大的话,比如下图横轴0-1,纵轴0-10000。所以先进行标准化是必要的。
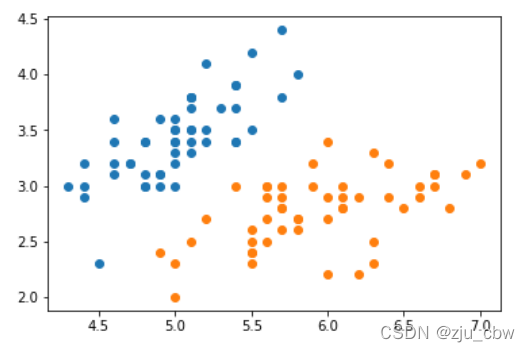
第一步,准备一个简单二分类数据集:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
| import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
iris = datasets.load_iris()
x = iris.data
y = iris.target
x = x[y<2, :2]
y = y[y<2]
plt.scatter(x[y==0, 0], x[y==0, 1])
plt.scatter(x[y==1, 0], x[y==1, 1])
plt.show()
|
第二步,实现svm,先使用一个比较大的C。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
|
from sklearn.preprocessing import StandardScaler
from sklearn.svm import LinearSVC
standardscaler = StandardScaler()
standardscaler.fit(x)
x_standard = standardscaler.transform(x)
svc = LinearSVC(C=1e9)
svc.fit(x_standard, y)
def plot_decision_boundary(model, axis):
x0, x1 = np.meshgrid(np.linspace(axis[0], axis[1], int((axis[1] - axis[0])*100)).reshape(1, -1),
np.linspace(axis[2], axis[3], int((axis[3] - axis[2])*100)).reshape(1, -1),)
x_new = np.c_[x0.ravel(), x1.ravel()]
y_predict = model.predict(x_new)
zz = y_predict.reshape(x0.shape)
from matplotlib.colors import ListedColormap
custom_cmap = ListedColormap(['#EF9A9A', '#FFF59D', '#90CAF9'])
plt.contourf(x0, x1, zz, linewidth=5, cmap=custom_cmap)
plot_decision_boundary(svc, axis=[-3, 3, -3, 3])
plt.scatter(x_standard[y==0, 0], x_standard[y==0, 1], color='red')
plt.scatter(x_standard[y==1, 0], x_standard[y==1, 1], color='blue')
plt.show()
|
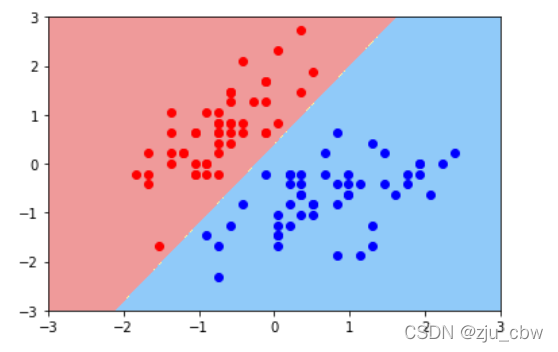
第三步:使用一个比较小的C,对比C取不同值的效果。
1
2
3
4
5
6
7
| svc2 = LinearSVC(C=0.01)
svc2.fit(x_standard, y)
plot_decision_boundary(svc2, axis=[-3, 3, -3, 3])
plt.scatter(x_standard[y==0, 0], x_standard[y==0, 1], color='red')
plt.scatter(x_standard[y==1, 0], x_standard[y==1, 1], color='blue')
plt.show()
|
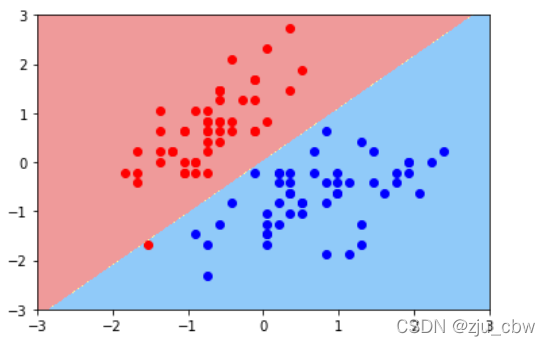
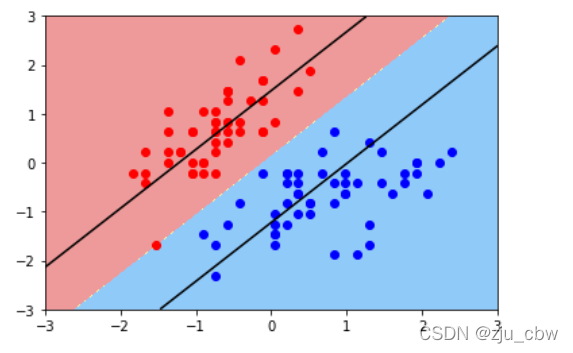
对比两幅图可以发现,当C较小时,误将一个红色的点分到蓝色当中,这也再次验证了当C越小,就意味着有更大的容错空间。
第四步:查看线性SVM的截距和系数
1
2
3
4
| svc.coef_
svc.intercept_
|
第五步:画出除了决策边界以外的两条跟支持向量相关的直线
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
| def plot_svc_decision_boundary(model, axis):
x0, x1 = np.meshgrid(np.linspace(axis[0], axis[1], int((axis[1] - axis[0])*100)).reshape(1, -1),
np.linspace(axis[2], axis[3], int((axis[3] - axis[2])*100)).reshape(1, -1),)
x_new = np.c_[x0.ravel(), x1.ravel()]
y_predict = model.predict(x_new)
zz = y_predict.reshape(x0.shape)
from matplotlib.colors import ListedColormap
custom_cmap = ListedColormap(['#EF9A9A', '#FFF59D', '#90CAF9'])
plt.contourf(x0, x1, zz, linewidth=5, cmap=custom_cmap)
w = model.coef_[0]
b = model.intercept_[0]
plot_x = np.linspace(axis[0], axis[1], 200)
up_y = -w[0]/w[1] * plot_x - b/w[1] + 1/w[1]
down_y = -w[0]/w[1] * plot_x - b/w[1] - 1/w[1]
up_index = (up_y >= axis[2]) & (up_y <= axis[3])
down_index = (down_y >= axis[2]) & (down_y <= axis[3])
plt.plot(plot_x[up_index], up_y[up_index], color='black')
plt.plot(plot_x[down_index], down_y[down_index], color='black')
plot_svc_decision_boundary(svc, axis=[-3, 3, -3, 3])
plt.scatter(x_standard[y==0, 0], x_standard[y==0, 1], color='red')
plt.scatter(x_standard[y==1, 0], x_standard[y==1, 1], color='blue')
plt.show()
|
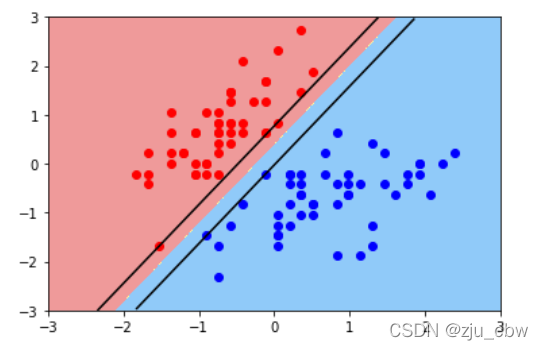
1
2
3
4
| plot_svc_decision_boundary(svc2, axis=[-3, 3, -3, 3])
plt.scatter(x_standard[y==0, 0], x_standard[y==0, 1], color='red')
plt.scatter(x_standard[y==1, 0], x_standard[y==1, 1], color='blue')
plt.show()
|
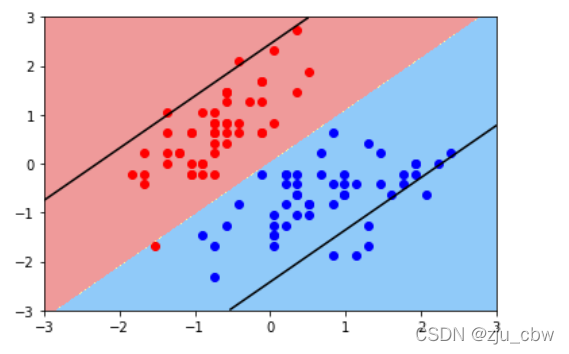
1
2
3
4
5
6
7
8
9
10
11
| svc3 = LinearSVC(C=0.1)
svc3.fit(x_standard, y)
plot_svc_decision_boundary(svc3, axis=[-3, 3, -3, 3])
plt.scatter(x_standard[y==0, 0], x_standard[y==0, 1], color='red')
plt.scatter(x_standard[y==1, 0], x_standard[y==1, 1], color='blue')
plt.show()
|
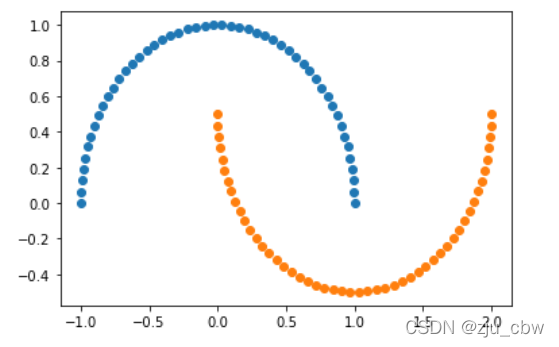
SVM中使用多项式特征
前面一直都在讲的是线性的svm,对于svm来说也可以解决非线性问题,类比线性回归到非线性回归的思想,首先使用多项式特征。
首先生成数据集:
1
2
3
4
5
6
7
8
9
10
11
12
| import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
x, y = datasets.make_moons()
x.shape
y.shape
plt.scatter(x[y==0, 0], x[y==0, 1])
plt.scatter(x[y==1, 0], x[y==1, 1])
plt.show()
|
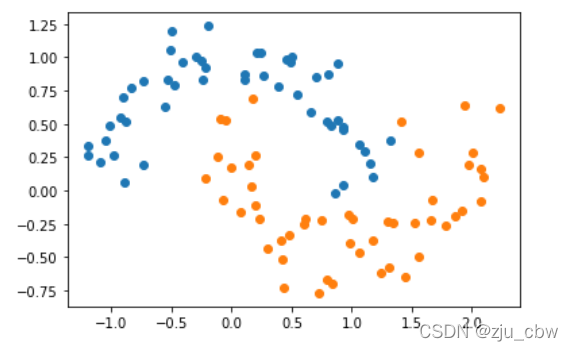
接下来给数据添加一些随机噪声:
1
2
3
4
| x, y = datasets.make_moons(noise=0.15, random_state=666)
plt.scatter(x[y==0, 0], x[y==0, 1])
plt.scatter(x[y==1, 0], x[y==1, 1])
plt.show()
|
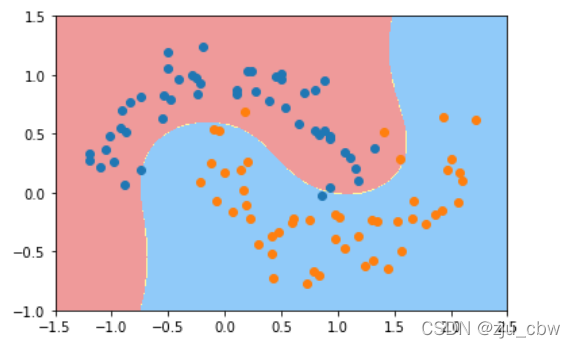
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
| from sklearn.preprocessing import PolynomialFeatures, StandardScaler
from sklearn.svm import LinearSVC
from sklearn.pipeline import Pipeline
def PolynomiaSVC(degree, C=1.0):
return Pipeline([
('poly', PolynomialFeatures(degree=degree)),
('std_scale', StandardScaler()),
('linear_svc', LinearSVC(C=C))
])
poly_svc = PolynomiaSVC(degree=3)
poly_svc.fit(x, y)
def plot_decision_boundary(model, axis):
x0, x1 = np.meshgrid(np.linspace(axis[0], axis[1], int((axis[1] - axis[0])*100)).reshape(1, -1),
np.linspace(axis[2], axis[3], int((axis[3] - axis[2])*100)).reshape(1, -1),)
x_new = np.c_[x0.ravel(), x1.ravel()]
y_predict = model.predict(x_new)
zz = y_predict.reshape(x0.shape)
from matplotlib.colors import ListedColormap
custom_cmap = ListedColormap(['#EF9A9A', '#FFF59D', '#90CAF9'])
plt.contourf(x0, x1, zz, linewidth=5, cmap=custom_cmap)
plot_decision_boundary(poly_svc, axis=[-1.5, 2.5, -1.0, 1.5])
plt.scatter(x1[y1==0, 0], x1[y1==0, 1])
plt.scatter(x1[y1==1, 0], x1[y1==1, 1])
plt.show()
|
除了使用这种增加多项式特征之后再给入线性svc中之外,还有一种方法可以实现类似的功能。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
| from sklearn.svm import SVC
def PolynomiaKernelSVC(degree, C=1.0):
return Pipeline([
('std_scale', StandardScaler()),
('kernel_svc', SVC(kernel='poly', degree=degree, C=C))
])
poly_kernel_svc = PolynomiaKernelSVC(degree=3)
poly_kernel_svc.fit(x, y)
plot_decision_boundary(poly_svc, axis=[-1.5, 2.5, -1.0, 1.5])
plt.scatter(x[y==0, 0], x[y==0, 1])
plt.scatter(x[y==1, 0], x[y==1, 1])
plt.show()
|
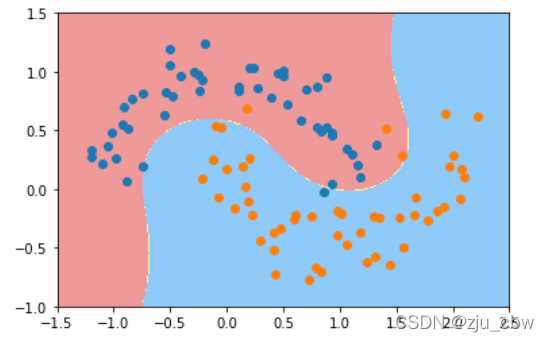
这种方法就是svm中kernel函数。接下来具体说明核函数。
核函数
在现实任务中,原始的样本空间也许并不存在一个能正确划分两类的超平面,对于这样一个问题,可将样本从原始空间映射到一个更高维的特征空间,使得样本在这个特征空间内线性可分。因此核函数的作用就是使得原本线性不可分的数据变得线性可分。下面是一个使用多项式核映射的过程。
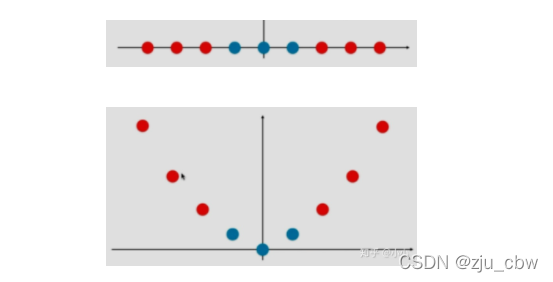
第一步:向高维空间映射

第二步:对偶问题的证明过程

第三步:经过对偶问题的求解后即可得到,

常用核函数:

接下里重点介绍高斯核函数。
高斯核函数
高斯核函数的目的就是将每一个样本点映射到一个无穷维的特征空间。实质上就是把一个m*n维的数据映射成了m*m的数据。由于理论上数据量可以是无穷维,所以说是映射到一个无穷维空间中。
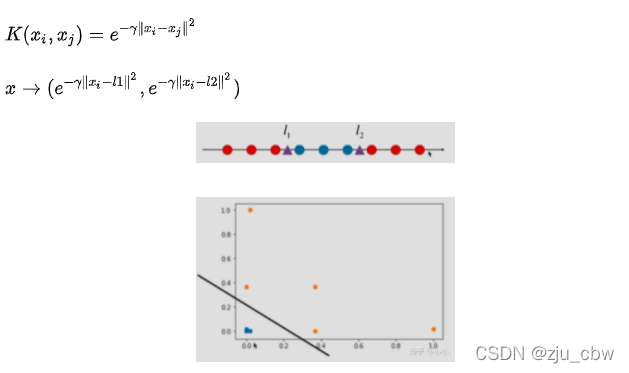
接下来通过高斯核函数映射来更加直观地理解整个映射的过程。
1
2
3
4
5
6
7
8
9
10
| import numpy as np
import matplotlib.pyplot as plt
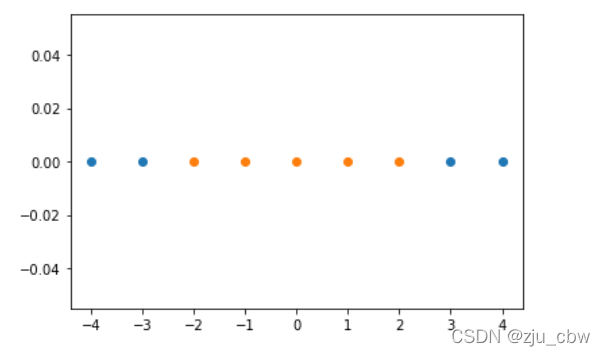
x = np.arange(-4, 5, 1)
y = np.array((x >= -2) & (x <= 2), dtype='int')
plt.scatter(x[y==0], [0] * len(x[y==0]))
plt.scatter(x[y==1], [0] * len(x[y==1]))
plt.show()
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
| def gaussian(x, l):
gamma = 1.0
return np.exp(-gamma *(x-l)**2)
l1, l2 = -1, 1
x_new = np.empty((len(x), 2))
for i,data in enumerate(x):
x_new[i, 0] = gaussian(data, l1)
x_new[i, 1] = gaussian(data, l2)
plt.scatter(x_new[y==0, 0], x_new[y==0, 1])
plt.scatter(x_new[y==1, 0], x_new[y==1, 1])
plt.show()
|
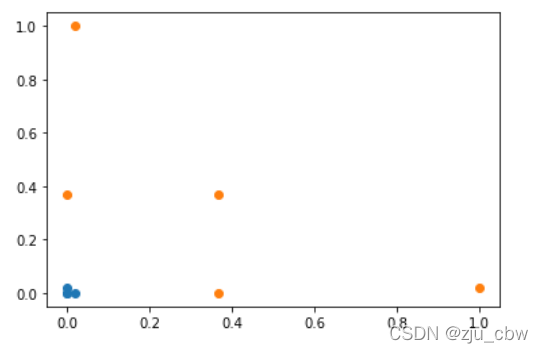
其实,真正的高斯核函数实现的过程中并不是固定的landmark,而是对于每一个数据点都是landmark。


接下来,使用sklearn中封装的高斯核函数:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
| import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
x, y = datasets.make_moons(noise=0.15, random_state=666)
from sklearn.preprocessing import StandardScaler
from sklearn.svm import SVC
from sklearn.pipeline import Pipeline
def RBFKernelSVC(gamma=1.0):
return Pipeline([
('std_scale', StandardScaler()),
('svc', SVC(kernel='rbf', gamma=gamma))
])
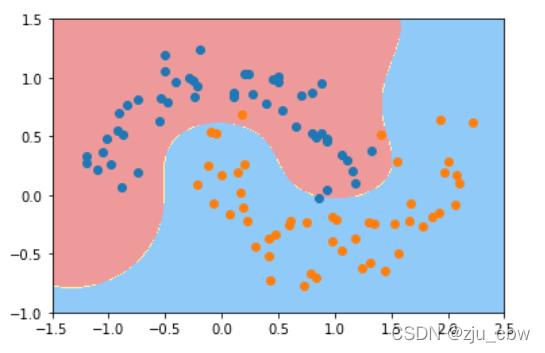
svc = RBFKernelSVC(gamma=1.0)
svc.fit(x, y)
def plot_decision_boundary(model, axis):
x0, x1 = np.meshgrid(np.linspace(axis[0], axis[1], int((axis[1] - axis[0])*100)).reshape(1, -1),
np.linspace(axis[2], axis[3], int((axis[3] - axis[2])*100)).reshape(1, -1),)
x_new = np.c_[x0.ravel(), x1.ravel()]
y_predict = model.predict(x_new)
zz = y_predict.reshape(x0.shape)
from matplotlib.colors import ListedColormap
custom_cmap = ListedColormap(['#EF9A9A', '#FFF59D', '#90CAF9'])
plt.contourf(x0, x1, zz, linewidth=5, cmap=custom_cmap)
plot_decision_boundary(svc, axis=[-1.5, 2.5, -1.0, 1.5])
plt.scatter(x[y==0, 0], x[y==0, 1])
plt.scatter(x[y==1, 0], x[y==1, 1])
plt.show()
|
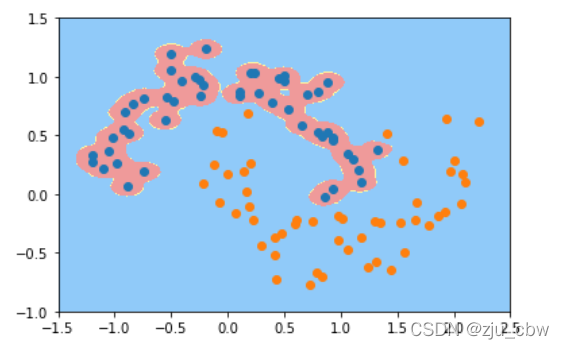
1
2
3
4
5
6
7
| svc_gamma100 = RBFKernelSVC(gamma=100)
svc_gamma100.fit(x, y)
plot_decision_boundary(svc_gamma100, axis=[-1.5, 2.5, -1.0, 1.5])
plt.scatter(x[y==0, 0], x[y==0, 1])
plt.scatter(x[y==1, 0], x[y==1, 1])
plt.show()
|
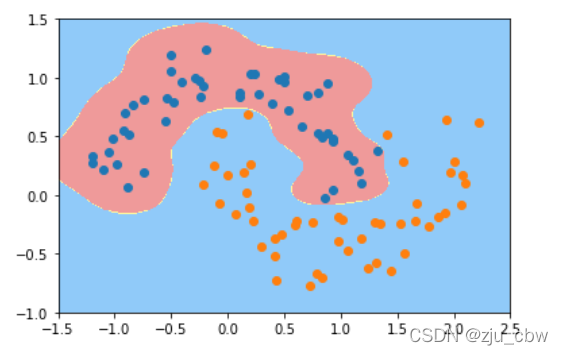
1
2
3
4
5
6
7
| svc_gamma10 = RBFKernelSVC(gamma=10)
svc_gamma10.fit(x, y)
plot_decision_boundary(svc_gamma10, axis=[-1.5, 2.5, -1.0, 1.5])
plt.scatter(x[y==0, 0], x[y==0, 1])
plt.scatter(x[y==1, 0], x[y==1, 1])
plt.show()
|
1
2
3
4
5
6
7
| svc_gamma01 = RBFKernelSVC(gamma=0.1)
svc_gamma01.fit(x, y)
plot_decision_boundary(svc_gamma01, axis=[-1.5, 2.5, -1.0, 1.5])
plt.scatter(x[y==0, 0], x[y==0, 1])
plt.scatter(x[y==1, 0], x[y==1, 1])
plt.show()
|
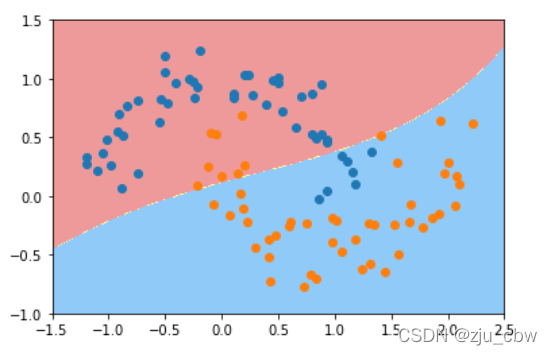
gamma相当于是在调节模型的复杂度,gammma越小模型复杂度越低,gamma越高模型复杂度越高。因此需要调节超参数gamma平衡过拟合和欠拟合。
SVM解决回归问题
svm解决回归问题的思路:在margin区域内的点越多越好。

在sklearn中实现SVM解决回归问题:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
| import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
boston = datasets.load_boston()
x = boston.data
y = boston.target
from sklearn.model_selection import train_test_split
x_train, x_test, y_train, y_test = train_test_split(x, y, random_state=666)
from sklearn.svm import SVR
from sklearn.svm import LinearSVR
from sklearn.preprocessing import StandardScaler
from sklearn.pipeline import Pipeline
def StandardLinearSVR(epsilon=0.1):
return Pipeline([
('std_scale', StandardScaler()),
('linear_svr', LinearSVR(epsilon=epsilon))
])
svr = StandardLinearSVR()
svr.fit(x_train, y_train)
svr.score(x_test, y_test)
|





































